Forschergruppe erweckt prähistorische Moleküle wieder zum „Leben“
Auf der Grundlage Zehntausende Jahre alter, zerstückelter DNA haben Wissenschaftler das Genom einer Gruppe von bisher unbekannten Bakterien rekonstruiert und ein prähistorisches Molekül wiederhergestellt. Für dieses nun in Science veröffentlichte Ergebnis haben sich Forscher verschiedenster Fachbereiche – von Archäologie über Bioinformatik bis Chemie – zusammengeschlossen: Mit den dafür entwickelten bioinformatischen Methoden wollen sie nach neuen Wirkstoffen suchen.
Bakterien stellen eine Vielzahl spannender Chemikalien her, sogenannte Naturstoffe – darunter zahlreiche Antibiotika und ande-re therapeutische Wirkstoffe. Für die Wissenschaft sind sie eine der wichtigsten Quellen neuer Arzneimittel – bisher suchen Forschende aber nur in heute lebenden Bakterien danach. Doch da Bakterien die Erde seit mehr als drei Milliarden Jahren besiedeln, gibt es in mittlerweile ausgestorbenen Bakterien eine enorme Vielfalt an Naturstoffen mit therapeutischem Potenzial.
Mikroben sind die größten Chemiker der Natur, und durch die Untersuchung der Genome uralter Bakterien lassen sich möglicherweise neue Verwendungsmöglichkeiten für sehr alte Moleküle entdecken.
Bakterien-DNA enthält Baupläne für Enzyme
Um diese Quelle zu erschließen, haben sich Forscher um den Chemiker Pierre Stallforth, Abteilungsleiter am Leibniz-Institut für Naturstoffforschung und Infektionsbiologie (Leibniz-HKI), und die Archäogenetikerin Christina Warinner, Associate Professor an der Harvard University und Gruppenleiterin am Max-Planck-Institut für evolutionäre Anthropologie (MPI-EVA), zusammengeschlossen. „Es ist uns erstmals gelungen, Substanzen neu herzustellen, die vor hunderttausend Jahren von Bakterien produziert wurden – die Paläofurane“, sagt Stallforth, der eine Professur an der Friedrich-Schiller-Universität Jena innehat. Als Grundlage diente Bakterien-DNA: Diese enthält die Baupläne für Enzyme, die wiederum chemische Verbindungen zusammenbauen können. „Mit dieser Studie haben wir einen wichtigen Meilenstein erreicht, um die enorme genetische und chemische Vielfalt unserer mikrobiellen Vergangenheit aufzudecken“, ergänzt Warinner.

Anhand der alten DNA ist es den Biochemikern gelungen, Moleküle herzustellen – die Paläofurane (hier in Pulverform).
Wenn ein Organismus stirbt, wird seine DNA schnell abgebaut und zerfällt in eine Vielzahl winziger Bruchstücke. Wissenschaftler können einige dieser DNA-Fragmente identifizieren, indem sie sie mit Datenbanken heutiger Organismen abgleichen. Doch ein Großteil der DNA gehört zu unbekannten, heute möglicherweise ausgestorbenen Mikroorganismen.
Die jüngsten Fortschritte in der Informatik machen es jedoch möglich, die DNA-Fragmente wieder zusammenzusetzen – ähnlich wie die Teile eines Puzzles – um auch unbekannte Gene und Genome zu rekonstruieren. Bei den stark abgebauten, extrem kurzen DNA-Fragmenten aus der Steinzeit eine große Herausforderung: „Wir mussten unseren Ansatz völlig neu überdenken“, erklärt Alexander Hübner, Postdoktorand am MPI-EVA. Drei Jahre des Testens und Optimierens später, so Hübner, einer der Erstautoren der Studie, haben sie einen Durchbruch erzielt: Es gelang, DNA-Abschnitte mit einer Länge von mehr als 100.000 Basenpaaren zu rekonstruieren und eine Vielzahl alter Gene und Genome wiederherzustellen. „Wir können jetzt beginnen, Milliarden unbekannter alter DNA-Fragmente systematisch in lange verschollene bakterielle Genome aus der Steinzeit einzuordnen.“
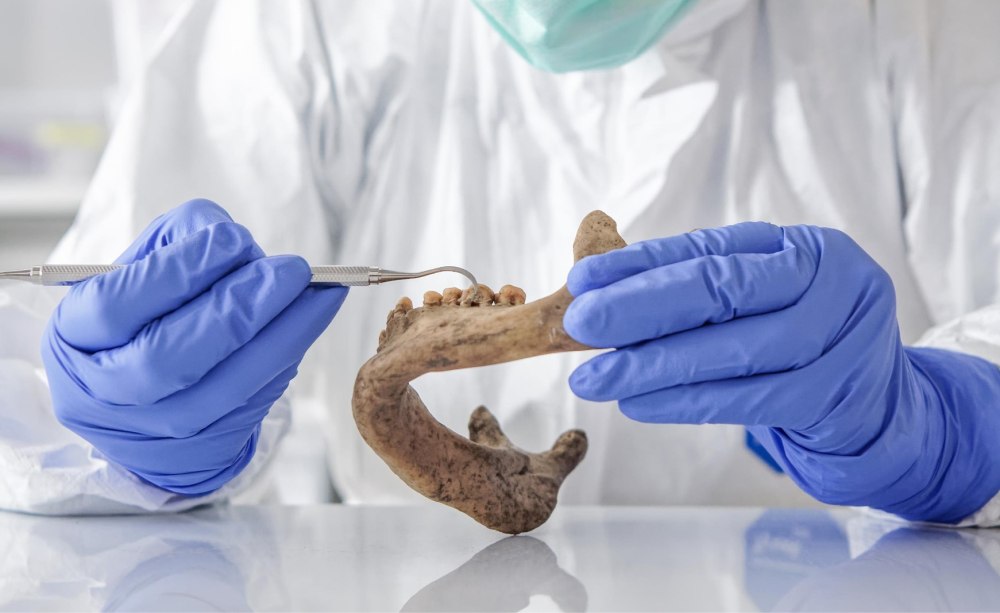
Um an die DNA von steinzeitlichen Mikroorganismen zu gelangen, nutzte das Team Zahnstein von Neandertalern, die vor etwa 100.000 bis 40.000 Jahren lebten, und von Menschen, die vor 30.000 bis 150 Jahren lebten.

Zahnstein konserviert die DNA über Jahrtausende hinweg und enthüllt so bisher verborgene Informationen über die Artenvielfalt und die funktionellen Fähigkeiten alter Mikroben.
Zahnbelag verwandelt in Friedhof mineralisierter Bakterien
Zahnstein ist der einzige Bestandteil des Körpers, der im Laufe des Lebens versteinert und lebenden Zahnbelag in einen Friedhof mineralisierter Bakterien verwandelt. Mithilfe modernster bioinformatischer Methoden rekonstruierten die Forschenden daraus die Genome zahlreicher Bakterienarten. „Die große bioinformatische Herausforderung lag darin, Fehler in der abgebauten DNA zu beheben und Verunreinigungen zum Beispiel durch jüngere DNA auszuschließen“, sagt Anan Ibrahim, Postdoktorandin am Leibniz-HKI und ebenfalls Erstautorin der Studie.
Neben vielen Bakterien, die auch heute noch die menschliche Mundflora besiedeln, fand sie ein unbekanntes Mitglied der Gattung Chlorobium. Dessen stark geschädigte DNA wies alle Merkmale eines fortgeschrittenen Alters auf und wurde im Zahnstein von sieben steinzeitlichen Menschen und Neandertalern gefunden. Alle sieben Chlorobium-Genome enthielten ein Biosynthese-Gencluster – den Bauplan für Enzyme – mit unbekannter Funktion. Ein besonders gut erhaltenes Chlorobium-Genom wurde aus dem Zahnstein der etwa 19.000 Jahre alten „Roten Dame von El Mirón“, Spanien, rekonstruiert. Das 2010 in einer spanischen Höhle gefundene Skelett ist der älteste Beleg für eine Beisetzung in der Epoche des Magdalénien auf der Iberischen Halbinsel.
„Nachdem wir diese rätselhaften alten Gene entdeckt hatten, wollten wir herauszufinden, was sie bewirken“, sagt Ibrahim. Mit modernsten biotechnologischen Methoden gelang es den Forschenden, die Gene in lebende Bakterien einzubauen, die daraus tatsächlich funktionale Enzyme bildeten. Damit sind sie die ersten, die diesen Ansatz erfolgreich auf Zehntausende Jahre alte Bakterien-DNA anwenden. Die reaktivierten Enzyme produzieren wiederum eine neue Familie mikrobieller Naturstoffe, die die Forschenden „Paläofurane“ genannt haben.
„Das ist der erste Schritt, um die verborgene chemische Vielfalt der Mikroben der Erdgeschichte zu erschließen“, sagt Martin Klapper, Postdoktorand am Leibniz-Institut für Naturstoffforschung und Infektionsbiologie und ein weiterer Erstautor der Studie.

Die Zusammenarbeit zwischen den Bereichen Paläogenomik und Chemie führt zu einem neuen Forschungsgebiet: der Paläobiotechnologie.
Neuer Forschungsbereich Paläobiotechnologie
Dieser Erfolg ist das direkte Ergebnis einer einzigartigen Zusammenarbeit von Forschenden aus Archäologie, Bioinformatik, Molekularbiologie und Chemie, die technologische und disziplinäre Barrieren überwinden und wissenschaftliches Neuland betreten wollten. „Mit der Förderung durch die Werner-Siemens-Stiftung wollen wir eine Brücke zwischen den Geistes- und Naturwissenschaften schlagen. Sie hat uns den Aufbau des neuen Forschungsbereichs Paläobiotechnologie ermöglicht“, sagt Stallforth.
Und Warinner ergänzt: „So konnten wir Technologien entwickeln, um Moleküle neu entstehen zu lassen, die bereits vor hunderttausend Jahren produziert wurden.“ Für die Zukunft hofft das Team, diese Herangehensweise zur Suche nach neuen Antibiotika einsetzen zu können.
Neben der Unterstützung durch die Werner-Siemens-Stiftung wurde die Arbeit unter anderem durch die Max-Planck-Gesellschaft, die Leibniz-Gemeinschaft und durch die Deutsche Forschungsgemeinschaft im Rahmen des Exzellenzclusters Balance of the Microverse und des Sonderforschungsbereichs ChemBioSys gefördert.
Originalpublikation online hier